目录
背景
- m6A修饰具有非常重要的功能
- 目前主要依赖于m6A-seq进行测定:受限于抗体,只有部分种类的修饰类型被测定到
- 使用三代测序,利用读段通过纳米孔产生的电流变化进行鉴定
- 电流变化需要比较电势图谱且重新map
- 开发基于三代测序数据进行m6A位点鉴定的方法
体外合成并测定
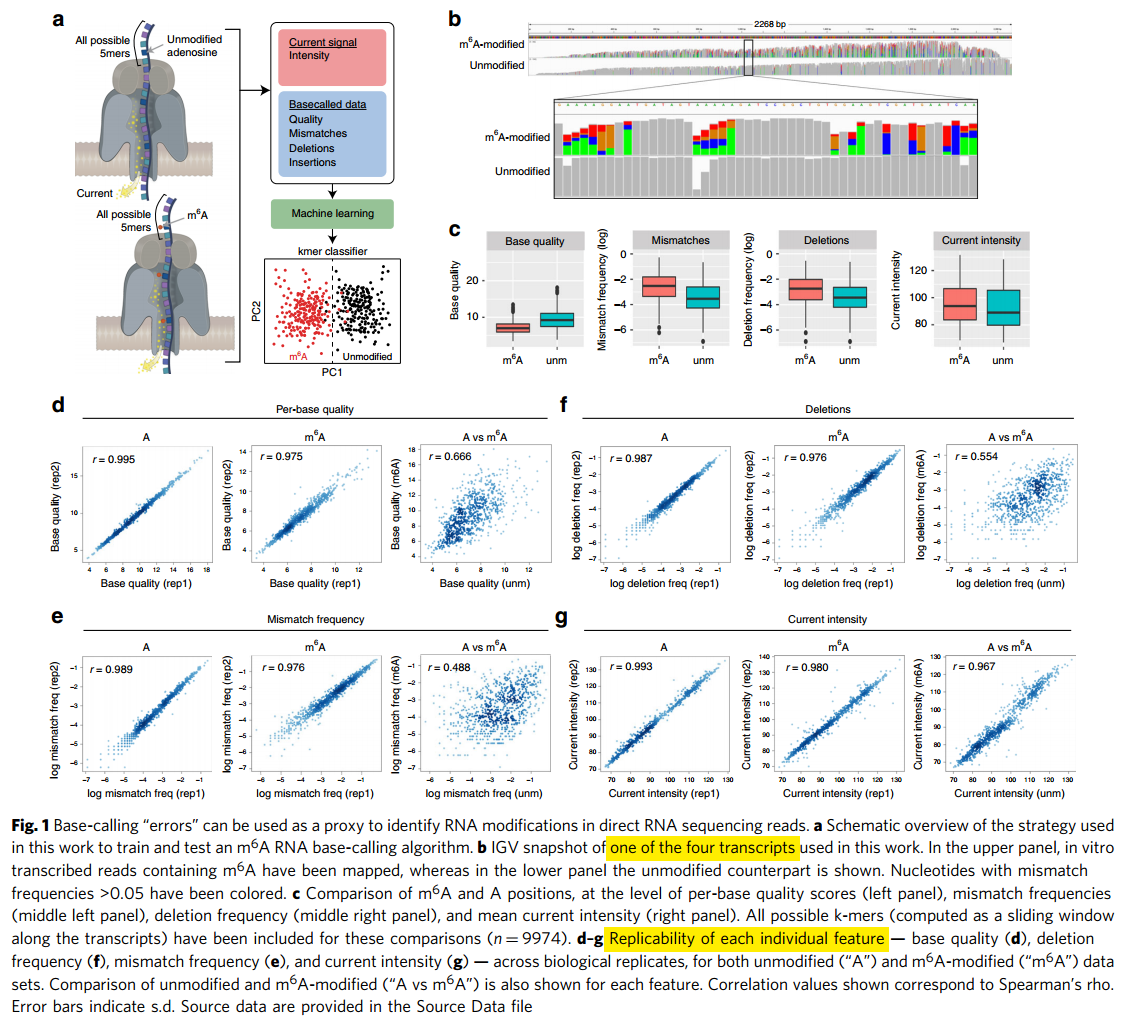
图解:首先是做了体外合成的实验
- a)体外合成的实验示意图。对于5bp长度的所有序列,每个碱基4个可能性,4^5=2^10=1024。对于合成的序列,分别测定有m6A修饰和没有修饰的序列,及此时电势(current)的大小。获得了对应条件下的特征:电势、碱基质量、错配频率、碱基删除和插入等,然后把这些特征进行机器学期模型的训练(这里是SVM)。
- 为什么选择5bp?m6A常规的motif序列是RRACH是5bp的。
- 做法:使用curlcake软件预测生成了1条10kb长的序列(包含了所有可能的1024个5mer,平均每个5mer出现10次),这里还考虑了预测的二级结构,使得二级结构最小。然后切成了四段,每段2000+长度,然后合成了这四条序列。
- b)其中一条合成序列的例子。在两个组中的track图,可以看到,在m6A修饰的组中,很多地方出现了突变(但是突变方向即突变后的碱基不固定),在对照组(相同序列但是没有m6A修饰的)就没有出现突变。
- c)在m6A组和对照组比较不同的指标的分布。具有区分性,且符合m6A修饰带来的测序质量更低、突变频率更高。
- d-g)不同指标在重复之间的相关性。这里没有列举出insertion,是因为这个指标在不同的rep之间重复性不好,因而不好作为m6A修饰预测时的特征。
基于测序得到的特征构建预测器
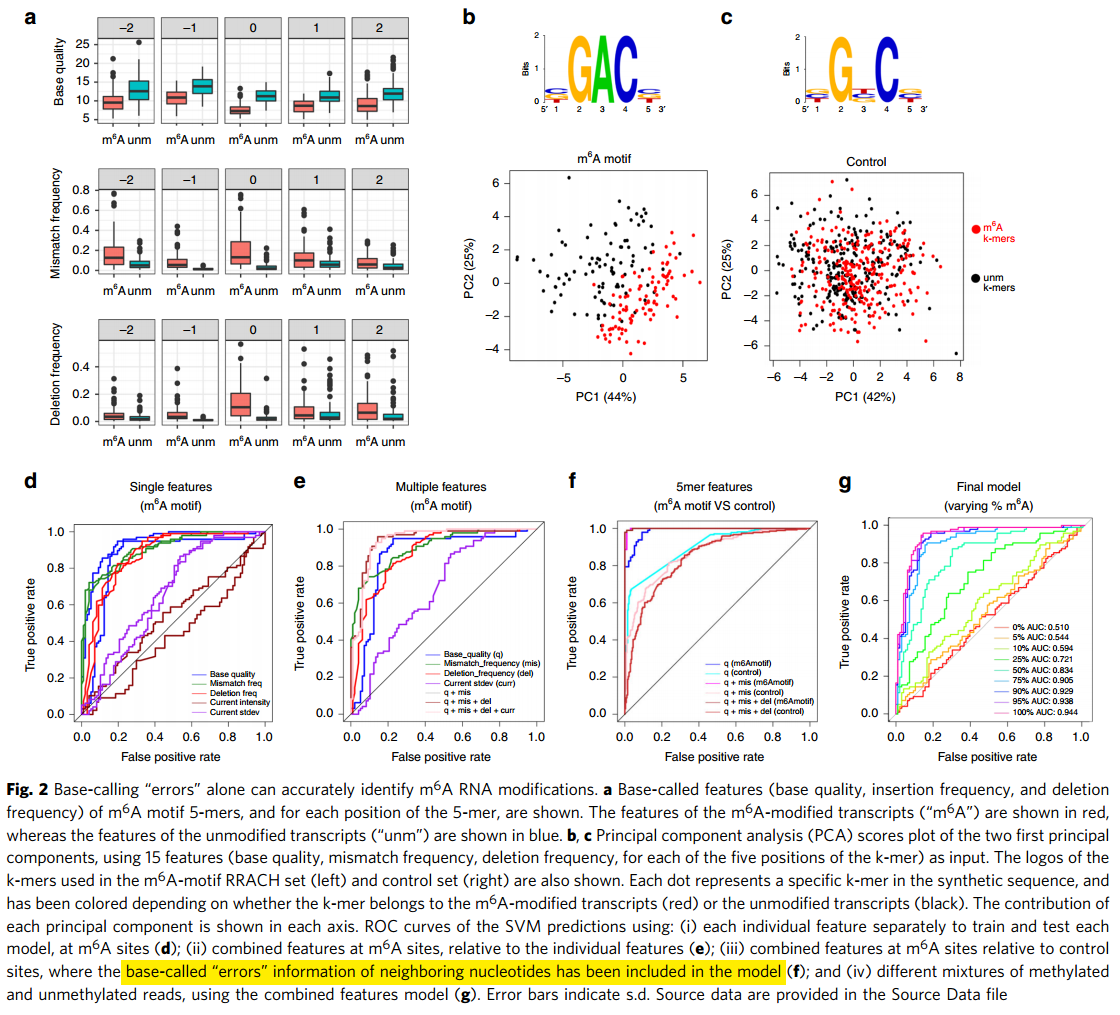
图解:
- a)每个碱基位置的特征分布,5bp x 3特征。上面图1是展示的所有可能的5mer的分布,这里展示的是m6A motif 5mer的分布,也是符合的,且每个碱基位点都是符合的。
- b-c)根据motif的feature是否可以区分m6A修饰的和没有修饰的?比如图b,有两套数据,一套是来源于含有m6A的合成序列,一个是来源于不含有m6A序列,但是都挑出具有m6A motif(RRACH)的5mer。现在根据3x5=15个特征进行PCA聚类,分成两类,发现分成的两类和他们的来源对应的很好,说明这些特征是具有很好的m6A区分性的(相同序列,但是特征不同,就可以判断m6A的修饰存在与否)。同理,挑出其他的非m6Amotif 5mer,做PCA,此时分不开,说明这些特征是特异的区分m6A的,而不是任何的都能区分开的。
- d)每个特征单独训练,是否能很好的预测m6A修饰?
- 70-86%的准确性。
- current的两个特征效果最差:43-65%准确性
- 训练集正样本:属于RRACH的5mer且来源于m6A合成序列,负样本:属于RRACH的5mer且来源于没有m6A修饰的合成序列。
- 这里为什么每个是两条线?两次的结果?那差异有点大啊
- e)使用不同的特征组合看效果,整体效果都好很多:88-91%准确性
- f)邻近的碱基特征使用进来,能提高预测的准确性:97-99%,但此时的假阳性也增高了,因此不应该使用其邻近位点的特征
- g)一定比例的修饰read和非修饰read混合,模型效果还行,即使25%的甲基化比例,能达到0.72的AUC。使用combined feature model预测。
预测体内的m6A修饰
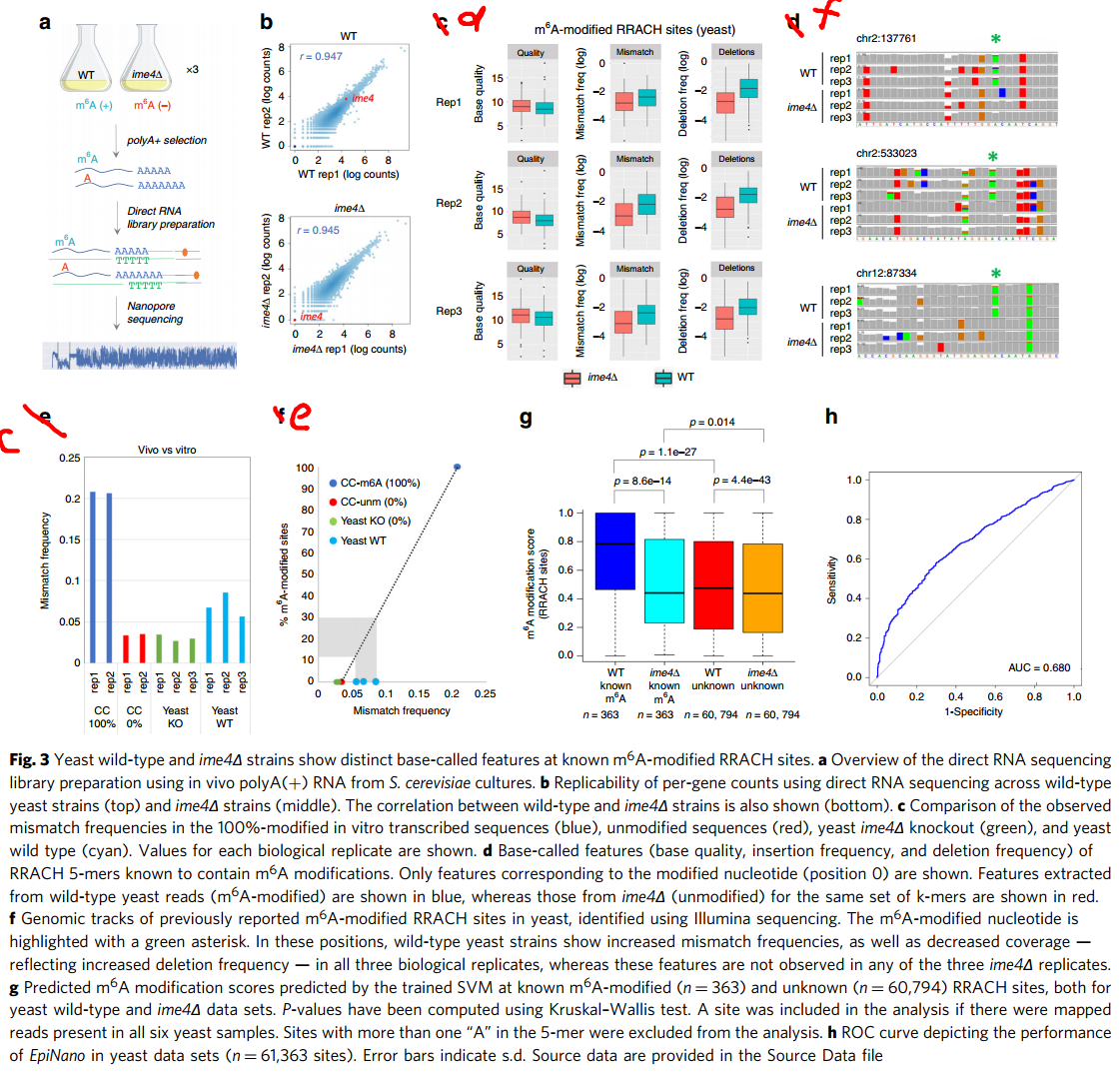
图解:这个图index和legend没有对应上。
- a)测定yeast数据。WT vs ime4 KO,后者就不能进行m6A修饰,相当于前面的control测定。分别测定,得到两个条件下的碱基特征值。
- b)read count在基因水平的重复之间相似性,同时把ime4的表达量label出来了。
- c)先看在以前鉴定的m6A位点上的特征分布。这里比较的是错配频率,可以看到,KO组比WT组低,KO组和之前invitro CC0%很接近,但是这里的WT组比100%CC也低了1倍。
- d)这是比较在以前鉴定的m6A位点上的几个特征在不同重复之间的分布,重复性很好,且符合修饰位点碱基质量更低、突变频率更高的特性。
- e)这里通过线性估计,在WT时,大概12-30%的m6A修饰,这个和之前的一个工作做得估计很接近。
- f)这是show的例子,但是有点懵!取了三个位点(之前其他工作已经鉴定过的),是在WT里面有明显突变的,但是ime4 KO没有。其他位点颜色呢?这样看:对于没有突变的位点,其整个bar是灰色的;如果有至少1个突变,则bar会显示各个碱基的比例。所有比如最右侧A,整个bar看起来是绿色的A,说明大部分是没有突变的,但是是存在其他突变碱基的。
- g)在WT和ime4 KO共有的RRACH位点是61163个,其中已知是m6A的有363个,未知的是609=794个。通过invitro得到的模型,预测这里invivo WT和ime4 KO数据里面m6A位点的score,然后看分布。
- WT known vs unknown:相同细胞系,已知和未知的,已知的预测得到分数更高,说明预测的很好
- ime4 KO known vs unknown:相同细胞系,已知和未知的,已知的预测得到分数接近,本身这些应该就是很接近的(都是没有m6A修饰的),说明预测的很好
- WT unknown vs ime4 KO unknown:有差异,说明存在潜在的新的m6A位点? h)在invivo数据达到0.68 AUC。
代码
相关的代码已经放在 EpiNano@github 上面了,可以参考。
参考
- Talk @London Calling 2019
- Eva Maria Novoa - Accurate detection of m6A RNA modifications in native RNA sequences using third-generation sequencing
- Accurate detection of m6A RNA modifications in native RNA sequences
If you link this blog, please refer to this page, thanks!
Post link:https://tsinghua-gongjing.github.io/posts/EpiNano.html
Previous:
使用迁移学习对scRNA数据降噪
Next:
AI在乳腺癌检测中的应用
Latest articles
Links
- ZhangLab , RISE database , THU life , THU info
- Data analysis: pandas , numpy , scipy
- ML/DL: sklearn , sklearn(中文) , pytorch
- Visualization: seaborn , matplotlib , gallery
- Github: me