目录
背景
- scRNA数据本身具有噪声及稀疏性
- 低表达量的转录本不能被很准确的定量
- 最近Nature method发表了一个方法SAVERX,通过对公共数据进行深度学习建模(autoencoder),然后使用迁移学习对靶标数据进行降噪
- 这里是对所有的表达量的数据的一个重新估计,不只是低丰度的或者没有值的,所以其功能不是imputation
模型
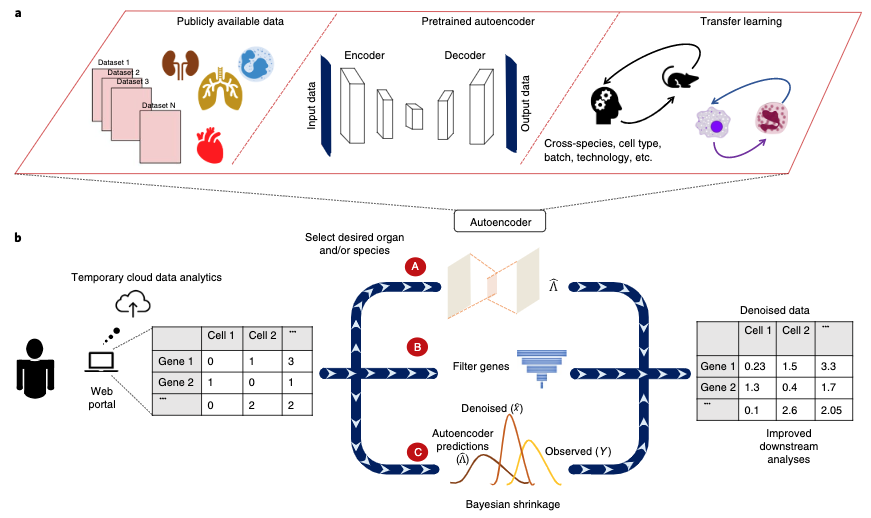
- 对于想要进行降噪处理的数据,分为3步进行
- (A)选择与靶标数据对应的数据,比如不同的物种、细胞系,通过autoencoder学习一个降噪的模型
- (B)通过cross-validation去除掉不能够预测的基因
- (C)使用贝叶斯模型估计最终的去噪声后的值
效果评估
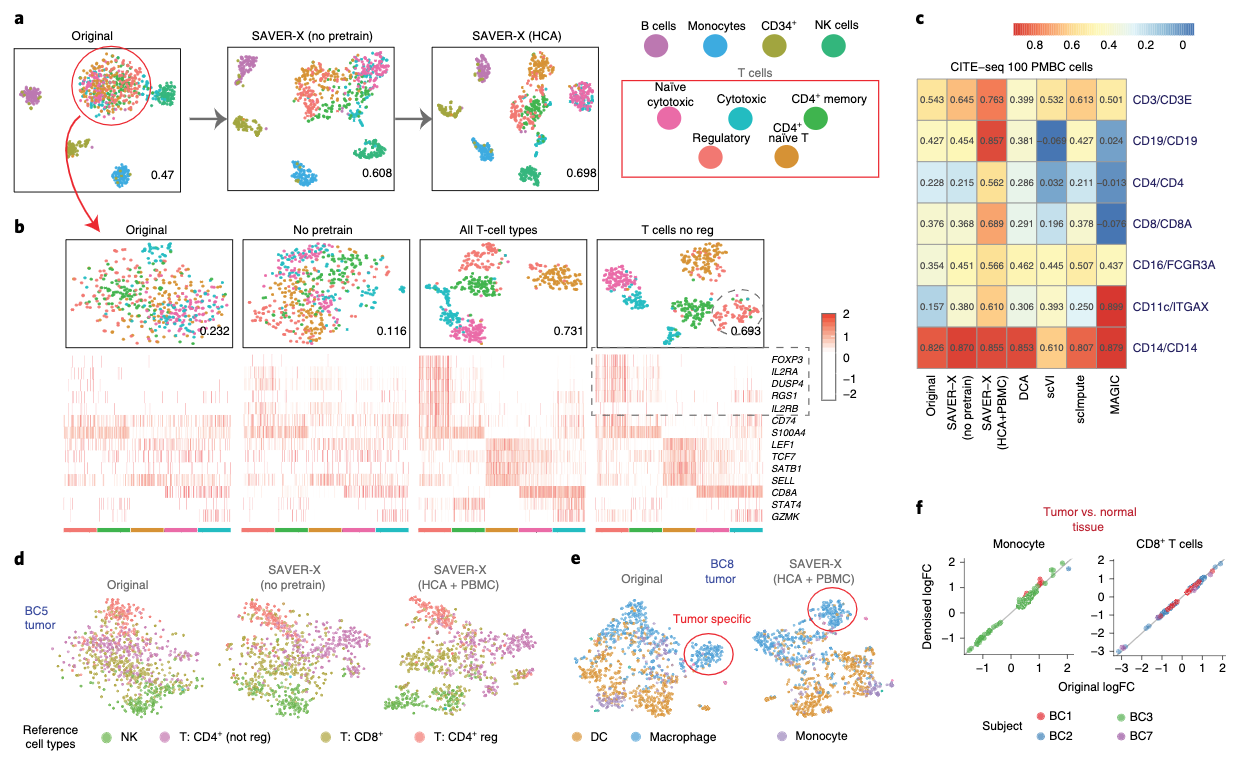
细胞系层次:先是在免疫细胞中进行了效果评估
- 可以看到,使用了迁移学习的经验知识后,聚类效果是更好的,也就是分的更开(图a)
- 一些marker基因也是的(图b)
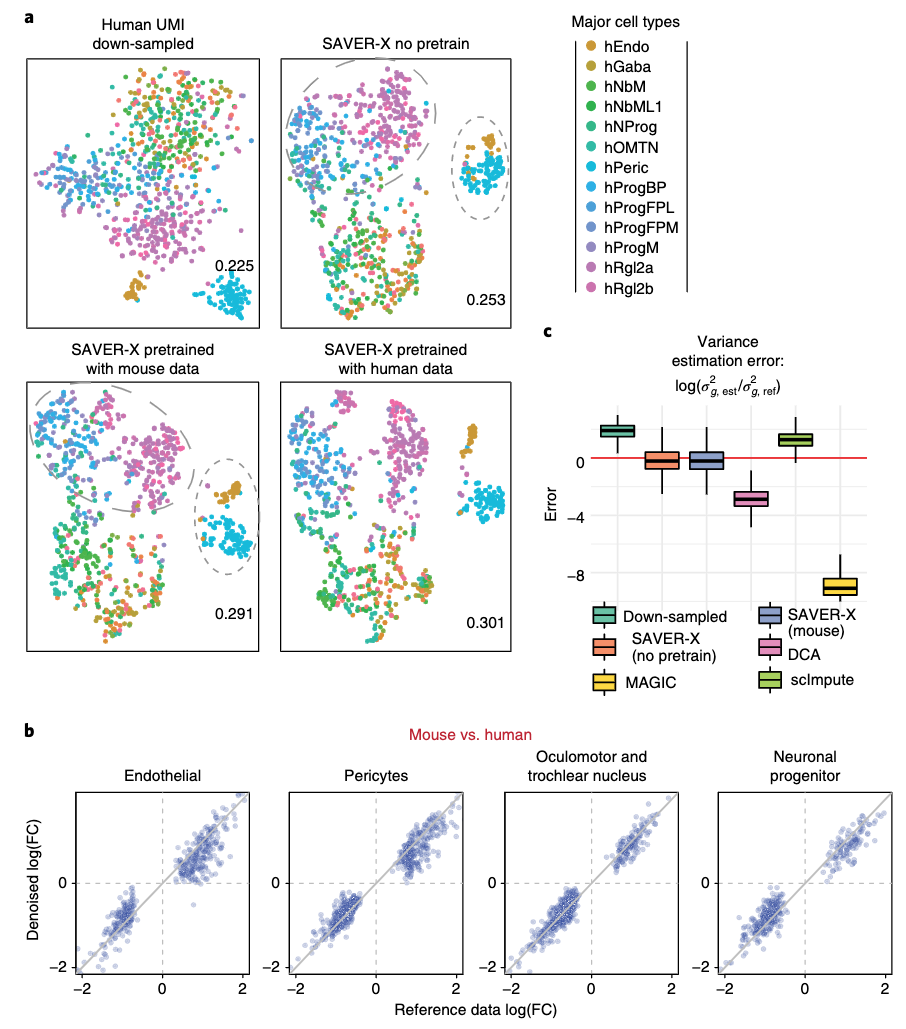
物种层次:人和鼠的迁移
- 从(a)可以看出,使用了已知数据学得的模型之后,聚类的效果更好了,不管是相同的物种的,还是其他物种的
If you link this blog, please refer to this page, thanks!
Post link:https://tsinghua-gongjing.github.io/posts/SAVER-X.html
Previous:
深度学习助力RNA可变剪切的预测
Next:
基于三代测序数据预测m6A修饰位点
Latest articles
Links
- ZhangLab , RISE database , THU life , THU info
- Data analysis: pandas , numpy , scipy
- ML/DL: sklearn , sklearn(中文) , pytorch
- Visualization: seaborn , matplotlib , gallery
- Github: me