14: Dimensionality Reduction
- 降维:非监督学习算法,用更简化的表示方法表征收集的数据。
- 为啥降维 -》压缩:
- 为啥降维 -》可视化:
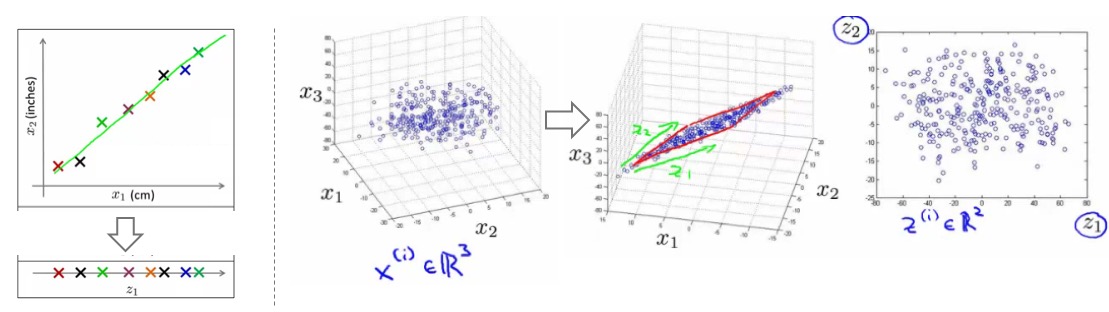
- 高维数据难以可视化
- 降维可以将数据以人可以理解的方式展现出来
- 比如一个表格,使用了50多个特征描述不同的国家,从这里我们能得到什么信息?
- 通常很难理解具体的降维之后的特征所表示的意思
- 主成分分析(PCA):
- 例子:2D数据降至1D,所以需要找一个一维的向量,使得所有点在这个向量的投射误差最小:
- 投映误差(projection error):点到向量的垂直距离(orthogonal distance),下图中的蓝色线条
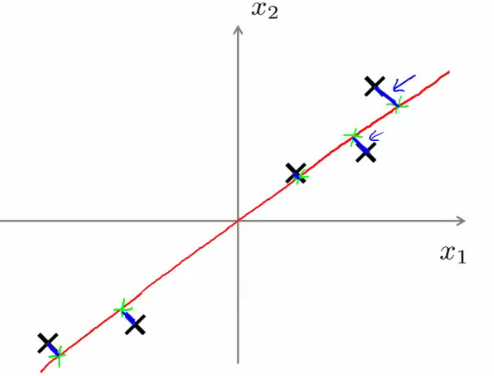
- 在做PCA之前,一定要对数据进行预处理:平均值归一化(mean normalization)和特征缩放(feature scaling)
- 广泛定义:
- nD -> kD,在将数据从
n维降至k维时,需要找k个向量,使得所有数据投射到这些向量时,投映误差最小。找的向量需要能最大程度的保留原来的信息,冗余的信息可以只保留部分信息,所以需要找的向量应该是波动最大(variance最大)的方向,这样也能保证投射误差最小。所以选取的k个方向,也是差异最大的k个方向。3D -> 2D:找两个向量。 - 特征向量:数据变化的方向向量。一个k维的数据,就有k个变化的方向,且不同的方向变化大小是不一样的。
- PCA vs 线性回归:
- PCA和线性回归很像,但PCA不是线性回归,其差别很大
- 线性回归:拟合直线,使得y方向的差值(vertical distance,预测的误差)最小,因为线性回归是有y值的;
- PCA:没有y值,只有特征值(且这些特征值都是同等对待的)。所以是找向量,误差的计算用的是垂直距离(orthogonal distance)。
- PCA算法:
- 【预处理:均值归一化】:对于每个特征,进行均值归一化,x -> x-mean(x)。每个特征的均值归一化到0.
- 【预处理:特征缩放】:当特征的区间差异很大时,需要进行缩放使得数值在可比较的区间里。1)Biggest - smallest;2)Standard deviation(标准差缩放更常见)
- 计算2个向量:1)新的平面向量u;2)新的特征向量z。
- 比如2D -> 1D,要计算向量u和新的特征向量:
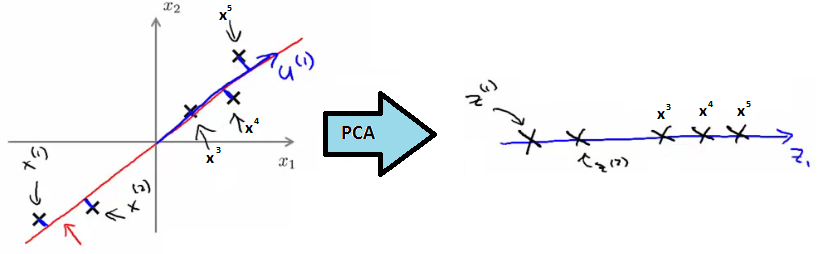
- 算法描述(这里有计算的步骤描述,可参考):
- 1)数据预处理:中心化,归一化。(对于图像、音频、文本等数据,通常不用做variance normalization归一化,比如特征乘以一个数,PCA计算的特征向量是一样的。)
- 2)根据样本计算协方差矩阵
- 3)利用特征值/奇异值分解,求特征值和特征向量
- 4)用特征向量构造投影矩阵
- 5)利用投影矩阵和样本数据,计算降维的数据
- 如何选取k:
- 通常会定义一个比值=平均投影误差/数据本身总方差:
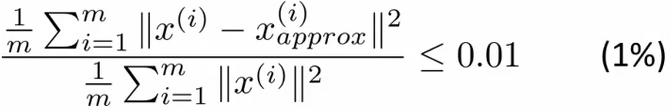
- 这个数值越小越好,越小说明丢失的信息越少,保留的信息越多。
- 实际操作:从k=1开始尝试,看哪个能满足上面的比值<=0.01,最小的能够满足这个比例的k值是我们想要的:

- 通过投影矩阵还原原始数据,我们只能用一个近似的原则。因为保留的k维是原始数据差异最大的,其他的维度差异近似为0,所以可以通过补充0的方式,来(近似)还原原始的数据:\(\begin{align} \hat{x} = U \begin{bmatrix} \tilde{x}_1 \\ \vdots \\ \tilde{x}_k \\ 0 \\ \vdots \\ 0 \end{bmatrix} = \sum_{i=1}^k u_i \tilde{x}_i. \end{align}\)
- 通常会定义一个比值=平均投影误差/数据本身总方差:
- PCA可加速监督式学习算法:
- 比如对于具有超高维特征的数据,可先用PCA进行降维,使用降维后的数据和原始的标签进行模型的训练,再去对于新的数据进行预测。
- 新数据:怎么预测?特征数目或者叫维度不一样!!!在上面的PCA中计算了参数投影矩阵。
- PCA应用:
- 压缩:减少存储,加速算法。保留多少的variance来确定k值。
- 可视化:通常降至2-3维
- 错误使用PCA避免过拟合:以为具有更少的特征就能避免过拟合,实则不然。应该使用正则化防止过拟合。
- 至少95%-99%的信息是需要保留的。
- 在使用PCA之前,先尝试不使用时能否达到预期的效果。
- 白化(whitening):目的是降低特征之间的相关性,特征的方差归一化
- 训练图片,相邻的像素是高度冗余的(特征具有强相关性)
- 通常学习算法希望的特征是:1)不相关的;2)具有相同的variance(彼此方差接近)。
- PCA-whitening:
- 是基于PCA的,包括两个步骤
- 1)使用PCA进行降维,消除特征之间的相关性:\(\textstyle x_{\rm rot}^{(i)} = U^Tx^{(i)}\)
- 2)方差归一化:\(\begin{align} x_{\rm PCAwhite,i} = \frac{x_{\rm rot,i} }{\sqrt{\lambda_i}}. \end{align}\)
- 这样就得到了白化的PCA,依然选取前k个作为主成分。
- ZCA-Whitening:
- 另外的一种白化方式,同样能够实现减少相关特征,方差归一化的目的。
- UFLDL笔记 - PCA and Whitening 一文较为详细的描述了PCA白化和ZCA变化的区别,可以参考一下。
- PCA白化是将原始数据投影到主成分轴上(消除特征相关性),然后对特征轴上的数据进行归一化(方差全缩放为1)。这些操作是在主成分空间完成的,为了使得白化的数据尽可能接近原始数据,所以可以把白化的数据转换回原始数据,这就是ZCA白化所做的事情。
- ZCA白化相当于将经过PCA白化后的数据重新变换回原来的空间。
If you link this blog, please refer to this page, thanks!
Post link:https://tsinghua-gongjing.github.io/posts/CS229-14-dimension-reduction.html
Latest articles
Links
- ZhangLab , RISE database , THU life , THU info
- Data analysis: pandas , numpy , scipy
- ML/DL: sklearn , sklearn(中文) , pytorch
- Visualization: seaborn , matplotlib , gallery
- Github: me