概述
支持向量机(Support Vector Machines):在分类与回归分析中分析数据的监督式学习模型与相关的学习算法。
- 原理:对于支持向量机来说,数据点被视为
p维向量,而我们想知道是否可以用p-1维超平面来分开这些点。这就是所谓的线性分类器。可能有许多超平面可以把数据分类。最佳超平面的一个合理选择是以最大间隔把两个类分开的超平面。 - 优点:泛化错误率低,计算开销不大,结果易解释
- 缺点:对参数和核函数的选取敏感
- 适用:数值型,标称型
知识点
- 线性可分(linearly separable):比如对于两组两种类别的数据,可用一条直线将两组数据点分开。
- 分隔超平面(separating hyperplane):将数据集分隔开来的直线。
- 数据点在二维,超平面就是一条直线;
- 数据点在三维,超平面就是一个平面;
- 数据点是1024维,超平面是1023维的对象。这个对象就叫做超平面(hyperplane)或者决策边界,位于超平面一侧的数据属于某个类别,另一侧的数据另一个类别。
- 间隔(margin):点到分隔面的距离。
- 支持向量(support vector):离分隔超平面最近的那些点。(所以到底是多少点算作?1个还是指定的任意个?)

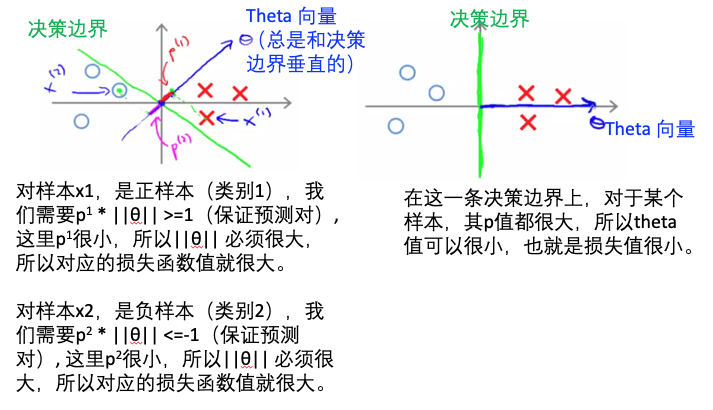
- 目标:构建分类器,数据点离超平面越远,则预测结果更可信。找到离分割面最近的点,确保他们离分割面越远越好。
- 寻找最大间隔:
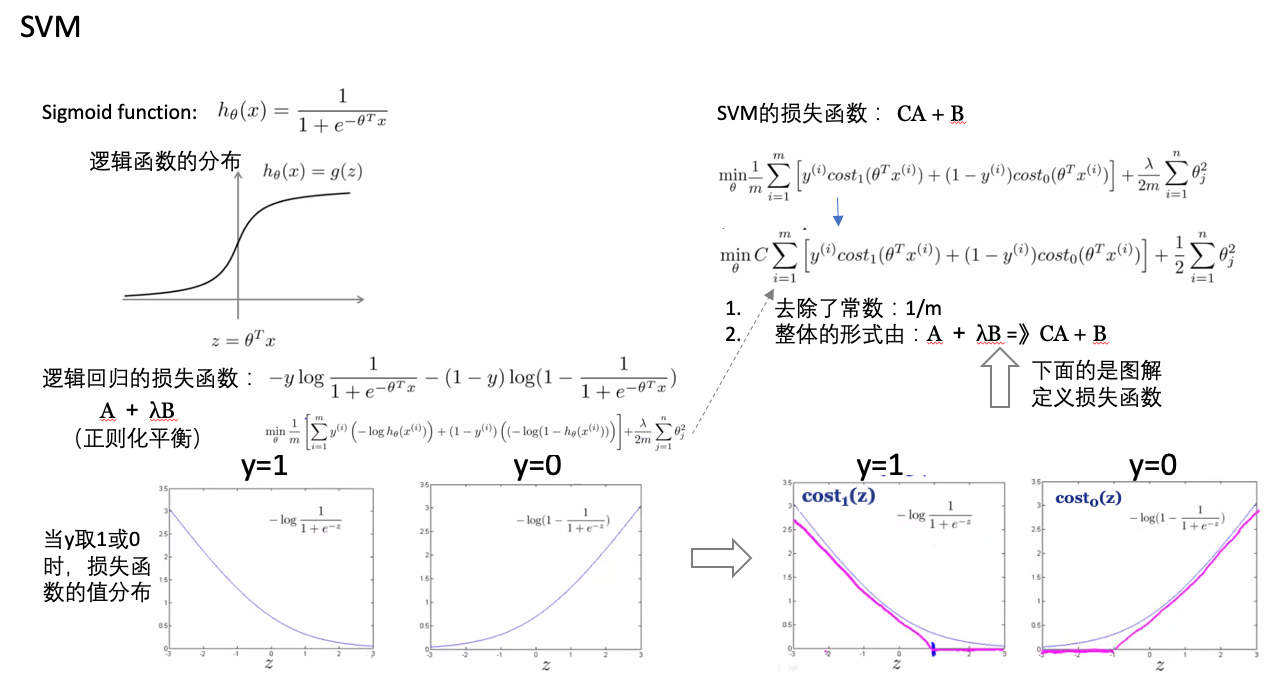
- 单位阶跃函数:f(x), 如果 x<0, f(x)=-1; 如果 x > 0, f(x)=1。类似于逻辑回归使用sigmoid函数进行映射,获得分类标签值。这里方便数学处理,所以输出是正负1,而不是0和1。
- 点到超平面的距离:WX+b
- 点到分割面的间隔:label *(WT+b),如果点在正向(+1,label为+1)且到超平面很远(正值),则此间隔值为一个很大的正数;如果点在负向(-1,label为-1)且到超平面很远(负值),则此间隔值也是一个很大的正数。
- 损失函数:
- SVM vs logistic regression:
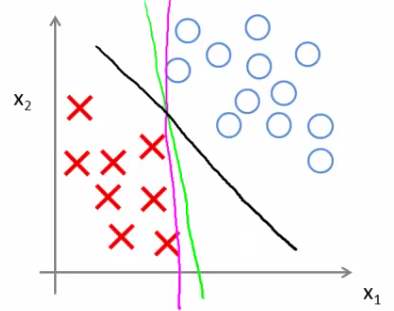
- 与逻辑回归相比,SVM是更稳健的,因为其选择的是最大距离(large margin),比如下图中逻辑回归选择了黄绿的边界,能够将现有数据分隔开,但是泛化能力差;黑色的是SVM选择的边界(最大化距离)。
实现
sklearn版本
用SVM预测肿瘤的类型:
#Import scikit-learn dataset library
from sklearn import datasets
#Load dataset
cancer = datasets.load_breast_cancer()
# print the names of the 13 features
print("Features: ", cancer.feature_names)
# print the label type of cancer('malignant' 'benign')
print("Labels: ", cancer.target_names)
# Import train_test_split function
from sklearn.model_selection import train_test_split
# Split dataset into training set and test set
X_train, X_test, y_train, y_test = train_test_split(cancer.data, cancer.target, test_size=0.3,random_state=109) # 70% training and 30% test
#Import svm model
from sklearn import svm
#Create a svm Classifier
clf = svm.SVC(kernel='linear') # Linear Kernel
#Train the model using the training sets
clf.fit(X_train, y_train)
#Predict the response for test dataset
y_pred = clf.predict(X_test)
#Import scikit-learn metrics module for accuracy calculation
from sklearn import metrics
# Model Accuracy: how often is the classifier correct?
print("Accuracy:",metrics.accuracy_score(y_test, y_pred))
参考
If you link this blog, please refer to this page, thanks!
Post link:https://tsinghua-gongjing.github.io/posts/SVM.html
Previous:
Python common used tricks
Latest articles
Links
- ZhangLab , RISE database , THU life , THU info
- Data analysis: pandas , numpy , scipy
- ML/DL: sklearn , sklearn(中文) , pytorch
- Visualization: seaborn , matplotlib , gallery
- Github: me